前言
自新冠疫情以来,mRNA技术因其在疫苗开发中的卓越表现而成为全球关注的焦点。从快速应对传染病到开发癌症疫苗和治疗遗传性疾病,mRNA展现了颠覆传统医疗的巨大潜力。而在这背后,5'端帽结构(Cap)作为mRNA的关键组成部分,不仅是翻译效率的“启动钥匙”,还在分子稳定性中扮演重要角色。加帽率作为mRNA质量的核心指标,直接影响其功能和临床应用,必须通过精准的检测方法进行全面表征。近日,伦敦帝国理工学院在《Current Opinion in Systems Biology》上发表综述[1],系统回顾了mRNA加帽率分析的最新技术。本文将深入解读这些方法的原理与进展,带您探索这一领域的科学前沿。
一、mRNA帽结构的角色与重要性
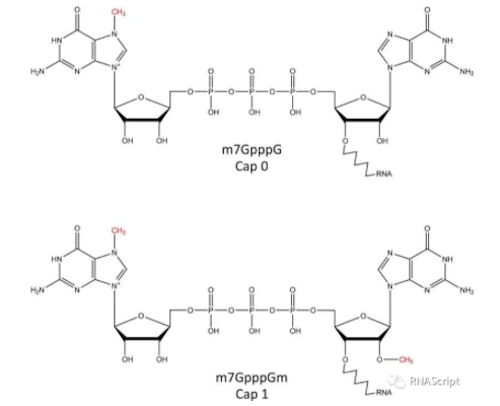
mRNA是一种单链核酸分子,通过转录DNA生成,负责将遗传信息转化为蛋白质。其5'端的帽结构(Cap)不仅是核糖体识别的信号,还通过与翻译起始因子eIF4E结合,启动蛋白质合成。此外,帽结构能有效阻断5'→3'核酸外切酶的降解作用,显著延长mRNA的体内半衰期。在体外转录(IVT)制备mRNA时,帽结构的引入尤为关键,目前常用的帽类似物包括ARCA(反向帽类似物)、Cap 0(m7GpppN)和Cap 1(m7GpppNm)。
加帽过程通常分为两种方式:
共转录加帽:在转录反应中直接加入帽类似物,操作简便。以Cap 1类似物为例,加帽率可达95%,但其合成受限于专利保护,且高纯度帽类似物的成本较高(每毫克价格可达数百美元)。
酶法加帽:利用牛痘加帽酶(Vaccinia Capping Enzyme, VCE)在IVT后分步添加帽结构,加帽率接近100%。然而,该方法需额外的酶促反应步骤(如2'-O-甲基转移酶处理),增加了质控复杂度和成本(酶成本约占总费用的30-50%)。
研究表明,未加帽的mRNA不仅翻译效率低下,还可能因触发RIG-I等免疫传感器而引发非特异性免疫反应。因此,加帽率的高低直接关系到mRNA疫苗或药物的安全性与疗效,精准检测成为研发和生产不可或缺的一环。
图片1:不同帽类似物的化学结构
二、mRNA加帽率检测方法
检测mRNA加帽率的核心难点在于:全长mRNA包含数千核苷酸,而加帽与未加帽的差异仅为一个甲基化鸟苷(m7G),其质量差异仅约0.01%。为克服这一挑战,科学家们开发了多种检测手段,从传统分离到高分辨率分析,以下是详细解读。
|
方法 |
灵敏度 (检测限) |
定量精度(CV/RSD) |
操作时间 |
高通量适用性 |
优点 |
缺点 |
|
电泳法 |
10 ng |
10-15% |
~5小时 |
低(需自动化) |
成本低、设备简单 |
耗时长、分辨率低 |
|
荧光帽类似物 |
1 pmol |
未定量(高背景) |
<1小时 |
中(快速筛选) |
操作直观、快速 |
背景干扰、标记效率低 |
|
qRT-PCR法 |
10^6-10^8拷贝 |
5-10% |
2-3小时 |
低(步骤多) |
定量精准、可特异性分析 |
步骤繁琐、酶依赖性高 |
|
LC-MS法 |
0.1 pmol |
<2% |
1-2小时 |
中(批次检测) |
高分辨率、重现性强 |
设备昂贵、需专业操作 |
|
纳米孔测序法 |
单分子级别 |
0.996准确率 |
~10分钟 |
中(需数据处理) |
无需酶切、高分辨率 |
需复杂数据分析 |
|
光学生物传感器 |
100 ng |
未精确定量 |
~30分钟 |
高(96/384孔板) |
高通量、操作简单 |
长链mRNA灵敏度低 |
表1 mRNA加帽率检测技术汇总
2.1 电泳法:简单但需优化
电泳法通过RNase H酶切mRNA,生成5'端短片段(20-50 nt),再利用聚丙烯酰胺凝胶电泳分离加帽与未加帽片段。加帽片段因带m7G而略重,迁移速率稍慢。这种方法设备要求低(仅需凝胶电泳仪),成本约为每样品5-10美元,但处理时间长(约5小时),且分辨率有限(难以区分Cap 0与Cap 1)。近年来,微流控毛细管电泳(如Agilent Bioanalyzer)将分析时间缩短至1分钟,检测极限可达10 ng RNA,结合荧光染料(如SYBR Gold)可提高灵敏度。然而,其定量依赖图像分析软件,误差率约10-15%,高通量应用仍需自动化改进。
2.2 荧光帽类似物:直观却待完善
荧光法通过合成带荧光团的帽类似物(如FITC标记的m7GpppG),直接测量加帽mRNA的荧光强度。例如,Ohno等人利用牛痘加帽酶的底物宽容性,开发了荧光标记GTP,检测限低至1 pmol。但该方法受限于标记效率(约70-80%),且背景荧光可能掩盖弱信号。此外,荧光团的化学稳定性(如光漂白)也影响长期检测。未来,开发三核苷酸帽类似物(如m7GpppNmN)并优化荧光团位置,或将成为快速筛选的理想选择。
2.3 qRT-PCR法:定量但繁琐
qRT-PCR通过特异性寡核苷酸桥(如qSL-RT-PCR)连接未加帽mRNA的5'端单磷酸,再经逆转录和PCR扩增,定量未加帽比例。例如,Morales等人利用胞嘧啶与m7G的弱配对,设计双链DNA接头,检测加帽mRNA的Ct值变化。该方法灵敏度高(可检测10^6-10^8拷贝),适用于混合样品分析。但其依赖多步酶反应(如T4 RNA连接酶),每次操作需2-3小时,且酶活性波动可能导致结果偏差(CV约5-10%)。高通量应用需整合自动化液处理系统。
2.4 液相色谱-质谱(LC-MS)法:行业标杆
LC-MS通过RNase H或核酶切割mRNA 5'端(约50 nt),分离并分析片段(如m7GpppG、GpppG、pppG)。加帽率由m7GpppG峰面积与总峰面积之比计算。诺华研究表明,酶法加帽的Cap 0含量达98%,而ARCA共转录加帽初始仅54.2%,通过优化ARCA:GTP比率(8:1)提升至92.8%。Moderna团队进一步验证,结合Q-Exactive质谱仪,检测限可达0.1 pmol,分辨率区分Cap 0与Cap 1达99%。LC-MS设备成本高(约50-100万美元),但其高精度和高重现性(RSD<2%)使其成为批次放行的行业标准。
2.5 纳米孔测序法:前沿探索
纳米孔测序利用蛋白孔(如α-溶血素)检测RNA通过时的电流变化,直接识别m7G等修饰。Wang等人通过工程化纳米孔和机器学习算法,实现了0.996的准确率,检测单次样品仅需10分钟,适合分析m6A、m7G等多重修饰。相比LC-MS,其无需酶切,样品制备时间缩短50%以上。但数据分析需高性能计算支持(如GPU集群),且孔隙堵塞问题可能降低通量。未来,优化孔径和算法将推动其产业化。
2.6 光学生物传感器:高通量潜力
光学生物传感器通过工程蛋白B4E(eIF4E与β-内酰胺酶融合)检测加帽mRNA。样品经poly-dT固相捕获,B4E结合帽后在492 nm处产生信号。Moya-Ramirez等人验证,该方法在96孔板中检测100 ng RNA只需30分钟,通量达数百样品/天。但对于>2 kb的长链mRNA,灵敏度下降至50%,因大分子干扰蛋白结合。未来,优化识别域或引入纳米颗粒标记可提升其适用性。
三、方法的优缺点与未来方向
从20世纪90年代的同位素标记到如今的LC-MS与纳米孔测序,mRNA加帽率检测技术经历了从粗放到精确的演变。以下是主要方法的对比:
电泳法:成本低($5-10/样品),但耗时长、分辨率低。
荧光法:检测快速(<1小时),但背景干扰大(S/N比约3:1)。
qRT-PCR:灵敏度高(10^6拷贝),但步骤复杂(CV 5-10%)。
LC-MS:分辨率高(RSD<2%),但设备昂贵($50万+)。
纳米孔测序:流程简单(10分钟/样品),但需数据分析支持。
光学生物传感器:高通量(数百样品/天),但长链mRNA灵敏度低。
未来方向包括:
1. 高分辨率分离:开发针对Cap 0、Cap 1及中间产物的凝胶配方(如变性琼脂糖),提升电泳分辨率。
2. 高通量优化:整合微流控与生物传感器,单次分析通量提升至千级。
3. 成本控制:通过化学合成低成本荧光帽类似物,替代昂贵酶试剂。
4. 多功能检测:结合纳米孔测序与LC-MS,同时分析加帽率和序列修饰(如m6A)。
结语
mRNA加帽率检测是确保mRNA产品质量的核心环节。从电泳法的简易起步,到LC-MS的精密标杆,再到纳米孔测序的前沿突破,每种方法都在为mRNA技术的进步添砖加瓦。随着分辨率、通量和成本的持续优化,未来的检测技术将更高效地支持mRNA疫苗和治疗性药物的开发,助力精准医疗迈向新高度。





